CRISPR/Cas系统简介
前言
CRISPR/Cas系统是普遍存在于细菌和古菌中的一类古老的免疫系统,用于抵御防御外源遗传元件(例如噬菌体)入侵。通过对它们的研究,科学家们开发出了CRISPR/Cas相关的一系列强大的基因编辑工具,是生物技术领域的重大突破之一。
CRISPR/Cas系统
CRISPR/Cas系统是原核生物的一种天然免疫系统,当某些细菌遭到病毒入侵后,能够把病毒基因的一小段存储到自身DNA里,这一存储空间被称为CRISPR。病毒再次入侵时,细菌能根据存写的片段识别病毒,用“基因魔剪”Cas(CRISPR associate system)蛋白将病毒的DNA切断[1]。
◆CRISPR/Cas系统组成
CRISPR/Cas系统由两个部分:CRISPR基因序列与基因组成[2]。
●CRISPR基因序列
由前导序列(leader)、重复序列(repeat)和间隔序列(spacer)构成。
①前导序列:富含AT碱基,位于CRISPR基因上游,被认为是CRISPR序列的启动子。
②重复序列:长度约20–50bp碱基,包含5–7bp回文序列,转录产物可以形成发卡结构,用于稳定RNA的整体二级结构。
③间隔序列:即被细菌俘获的外源DNA序列,相当于细菌免疫系统的“黑名单”,当这些外源遗传物质再次入侵时,CRISPR/Cas系统就会予以精准打击。
●Cas基因
位于CRISPR基因附近或分散于基因组其他地方,该基因编码的蛋白均可与CRISPR序列区域共同发生作用,在防御过程中至关重要,目前已经发现了Cas1-Cas10等多种类型的Cas基因。
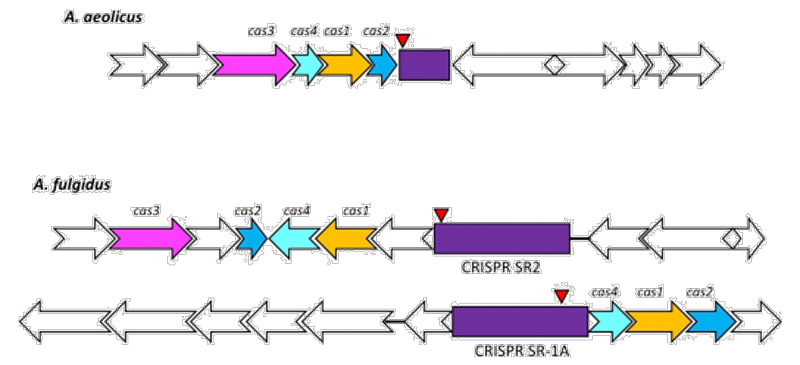
CRISPR基因座的基本结构[2]
CRISPR/Cas系统发现史
1987年,Ishino等首次在大肠埃希菌lap的上游序列区发现了14个约29bp的重复序列及32bp左右的间隔序列,之后又在化脓性链球菌和结核分枝杆菌中陆续发现相似序列[3]。
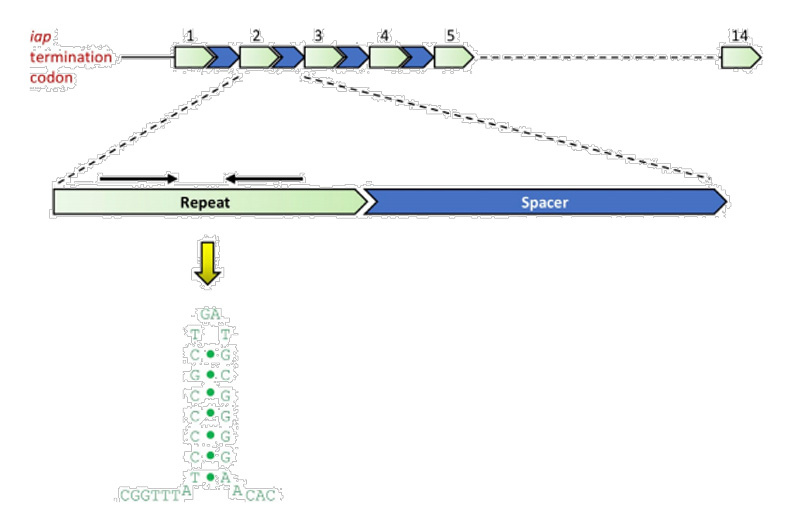
首次在大肠杆菌中发现CRISPR序列[2]
2000年Mojica等发现这些短重复序列存在于许多细菌和古细菌中,并将其命名为规则的短间隔重复序列(SRSRs)[4],2001年,She等进一步命名为大簇的20nt串联重复序列(LCTR)[5],2002年,Jansen等先后将这段序列命名为间隔的正向重复序列(SPIDR)和规律成簇的间隔短回文重复序列(CRISPR),同时将与其功能可能相关的4个基因(Cas1、Cas2、Cas3、Cas4)命名为Cas基因,且这种命名方式沿用至今[6-8]。
CRISPR/Cas系统分类
◆CRISPR/Cas系统分类方法的变更
2005年,Half等基于40多种细菌和真菌的分析结果,结合Cas1蛋白进化树的拓扑学分析以及Cas基因的操纵子序列信息等方面的差异,首次对CRISPR/Cas系统进行了分类,并将其分别归于8个基因组中,4个核心蛋白的名字沿用了2002年Jansen等的命名方式,而8个基因组中编码蛋白的基因命名由细菌来源和数字组成,如cse1(CRISPR system of E.coli gene number 1),这种分类方法虽然简单,但对于关系较远又有一定联系的蛋白分类命名并不科学[6-9]。
2011年,美国国立生物信息中心(NCBl) 提出了一种以Cas1、Cas2基因为系统核心的全新分类方式。Makarova等基于系统发育、基因组和结构分析的证据将现有的CRISPR/Cas系统分为Ⅰ型、Ⅱ型、Ⅲ型,大多数的CRISPR/Cas系统可归于3个类型之中,其余暂时无法分类的命名为U型,并将之前发现的部分基因进行了重新分类命名,如将cmx5重命名为Cas5,cmx6重命名为Cas6,在亚型的基础上进一步引用字母来避免一些同源基因无法被分类,如cse3和csy4被重命名为Cas6e和Cas6f[7,10]。
◆CRISPR/Cas系统现行分类方法
2015年,Makarova等对所有已知的93个CRISPR/Cas系统的蛋白家族进行分析并建立了一个包括394个位置特异性计分矩阵(PSSM)的书库,其中229个PSSMs是新增的,其利用这些矩阵搜索了2751个完整注释过细菌和真菌的基因组,预测出了1694个完整Cas基因的基因座,最终,1574个完整的基因座被分入先前已经分出的类型中或者新分类出的Ⅳ型、Ⅴ型系统中。经过分析总结,Makarova等认为应从更广泛、更基础的角度对CRISPR/Cas系统进行分类,即应将拥有多亚基crRNA效应复合物的归入一类,而少数蛋白亚基行使功能的被归入另一类,向下分类可以分出5个型,16个亚型,进而形成了现在应用的分类方法。即根据CRISPR/Cas系统干扰机制的不同,分为两大类(Class1和Class2),其中Class1由多个蛋白构成效应复合物(包括Ⅰ、Ⅲ和Ⅳ型);Class2由单个蛋白行使功能(包括Ⅱ、Ⅴ和Ⅵ型)[7,11]。
截至2019年3月1日,NCBI上共有13116个完整的古细菌和细菌的基因组,利用Blast技术可以鉴定到7915个Cas位点。截至2019年Makarova等关于CRISPR/Cas系统分类的研究中,亚型的分类数量达到了33个,较2015年增加了1倍,并且分出两个新的型,包括蛋白体型Ⅱ型、更小的Ⅴ型系统和以RNA为切割目标的Ⅵ型。目前来看,这种分类方法已经逐渐趋向于稳定,对CRISPR/Cas系统的分类也趋近于补充和完善的阶段[7,11]。
◆CRISPR/Cas系统包含的亚型
现在已知的CRISPR/Cas系统分为2个大类6个型,每个型又分为多个亚型,每个型包含的Cas蛋白也存在差异[7]。

CRISPR/Cas系统包含的亚型与蛋白[7]
CRISPR/Cas系统免疫机制
CRISPR/Cas系统发挥免疫作用主要经过3个阶段:适应阶段(Adaptation),表达阶段(Expression)和干扰阶段(Interference)[12,13]。
◆适应阶段
spacer捕获阶段,细菌识别出外源性DNA(病毒或者质粒),并且将其作为一个新的spacer整合到CRISPR位点前导序列的末端。
◆表达阶段
CRISPR RNA(crRNA)合成和表达的阶段,CRISPR位点转录出一个前体crRNA(pre-crRNA),随后合成成熟crRNAs,每个成熟的crRNA只包含一个spacer[12,13]。
◆干扰阶段
crRNA和Cas蛋白整合成一个蛋白效应复合物,利用crRNA识别和杀伤拥有与crRNA中spacer互补序列的任何噬菌体或质粒[12,13]。
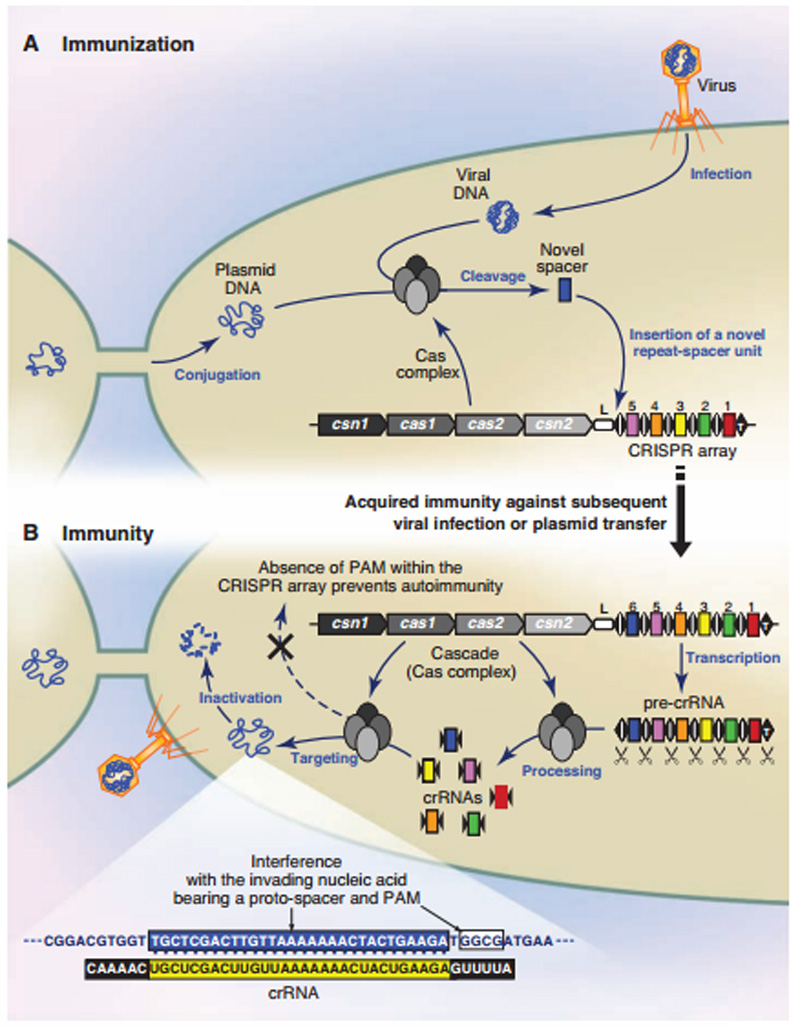
CRISPR/Cas免疫机制[13]
参考文献:
[1]周春燕 药立波. 生物化学与分子生物学 [M].北京: 人民卫生出版社, 2021.6.
[2]Agarwal N, Gupta R. History, evolution and classification of CRISPR-Cas associated systems. Prog Mol Biol Transl Sci. 2021;179:11-76.
[3]ISHINO Y, SHINAGAWAH, MAKINO K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozymeconversion in Escherichia coli, and identification of the gene productIJJ. J. Bacteriol., 1987, 169(12):5429-5433
[4]Mojica FJ, Díez-Villaseñor C, Soria E, Juez G. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria. Mol Microbiol. 2000 Apr;36(1):244-6.
[5]She Q, Singh RK, Confalonieri F, et al. The complete genome of the crenarchaeon Sulfolobus solfataricus P2. Proc Natl Acad Sci U S A. 2001 Jul 3;98(14):7835-40.
[6]Jansen R, van Embden JD, Gaastra W, et al. Identification of a novel family of sequence repeats among prokaryotes. OMICS. 2002;6(1):23-33.
[7]高维崧,窦金萍,韦双等.CRISPR/Cas系统的分类及研究现状[J].生物技术进展,2022,12(04):532-538.
[8]冯万有,蒙丽娜,陈春等.CRISPR/Cas系统分类和病原体检测的研究进展[J].生物技术,2022,32(02):258-267+251.
[9]Haft DH, Selengut J, Mongodin EF, et al. A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes. PLoS Comput Biol. 2005 Nov;1(6):e60.
[10]Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems. Nat Rev Microbiol. 2011 Jun;9(6):467-77.
[11]Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol. 2015 Nov;13(11):722-36.
[12]Hille F, Richter H, Wong SP, et al. The Biology of CRISPR-Cas: Backward and Forward. Cell. 2018;172(6):1239-1259.
[13]Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 2010;327(5962):167-170.








